理化学研究所
東京大学
京都大学
科学技術振興機構
理化学研究所(理研)開拓研究本部渡邉分子生理学研究室の渡邉力也主任研究員、篠田肇研究員、東京大学先端科学技術研究センターの西増弘志教授、同大学大学院理学系研究科の濡木理教授、京都大学ウイルス・再生医科学研究所の野田岳志教授らの共同研究グループ※は、新型コロナウイルス(SARS-CoV-2)由来のウイルスRNAを「1分子」レベルで識別して5分以内に検出する革新的技術の開発に成功しました。
本研究成果は、新型コロナウイルス感染症(COVID-19)などの超高感度・迅速診断装置の開発を含む、次世代の感染症診断法の核心技術としての応用展開が期待できます。
現在、新型コロナウイルスの確定診断には、ウイルスRNAを精製した後PCR法[1]などで増幅して検出するPCR検査が主に行われています。しかしPCR検査は、検出に最短で1時間程度かかり、検出エラーも発生することから、大量の検体を短時間かつ高精度に解析することが困難です。
今回、共同研究グループでは、世界最先端のマイクロチップ技術[2]と核酸検出技術CRISPR-Cas13a[3]を融合させることで、世界最速の新型コロナウイルスの検出法「CRISPR-based amplification-free digital RNA detection;SATORI)法」を開発しました。SATORI法を用いると、5分以内でウイルスRNAを1個ずつ識別して検出できます。検出感度は5フェムトモーラー(fM、fMは1000兆分の1モーラー)であり、新型コロナウイルス感染者の検体中のウイルスRNA量を検出する感度を満たしています。また、ランニングコストは9ドル程度と安価であるという利点もあります。
本研究は、科学雑誌『Communications Biology』のオンライン版(4月19日付:日本時間4月19日)に掲載されました。
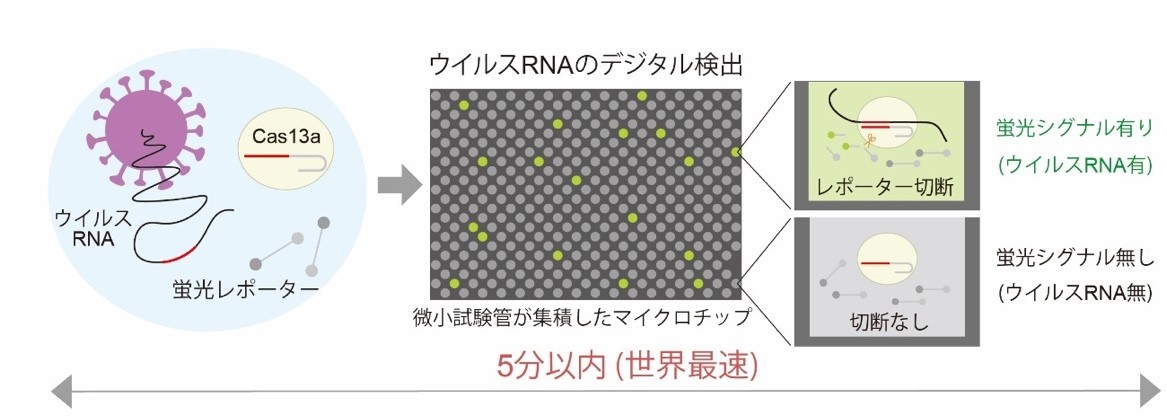
SATORI法によるウイルスRNAのデジタル検出原理
※共同研究グループ
理化学研究所 開拓研究本部 渡邉分子生理学研究室
主任研究員 渡邉 力也 (わたなべ りきや)
研究員 篠田 肇 (しのだ はじめ)
研究員 安藤 潤 (あんどう じゅん)
特別研究員(研究当時) 田口 裕也 (たぐち ゆうや)
テクニカルスタッフI 牧野 麻美 (まきの あさみ)
テクニカルスタッフII 高橋 千春 (たかはし ちはる)
東京大学 先端科学技術研究センター
教授 西増 弘志(にします ひろし)
学術専門職員 岡崎 早恵(おかざき さえ)
東京大学 大学院理学系研究科
教授 濡木 理 (ぬれき おさむ)
大学院生 中川 綾哉(なかがわ りょうや)
京都大学 ウイルス・再生医科学研究所
教授 野田 岳志(のだ たけし)
助教 中野 雅博(なかの まさひろ)
助教 村本 裕紀子(むらもと ゆきこ)
研究支援
本研究は、科学技術振興機構(JST)戦略的創造研究推進事業CREST「細胞外微粒子に起因する生命現象の解明とその制御に向けた基盤技術の創出(JPMJCR19H5)」、三菱財団自然科学研究特別助成「新型コロナウイルス等感染症に関する学術研究助成」 、日本医療研究開発機構(AMED)ウイルス等感染症対策技術開発事業(20he0622032h0001)、新興・再興感染症に対する革新的医薬品等開発推進研究事業(19fk0108113436、20fk0108270h0001)、JSPS科研費JP20H05931をはじめとする諸機関からの支援によって行われました。
1.背景
現在、新型コロナウイルス(SARS-CoV-2)の感染診断では、主にタンパク質抗原を検出する方法(抗原検査)とウイルスRNAを増幅して検出する方法(PCR検査)が利用されており、それぞれスクリーニング、確定診断など用途に応じて使い分けされています(図1)。
感染が疑われる場合、抗原検査を用いたスクリーニングが行われます。抗原検査は30分程度と迅速かつ簡便にウイルスを検出できるため、スクリーニングには適していますが、検出感度や特異度[4]の低さに起因する検出エラーの多さが問題となっています。一方、次のステージの確定診断として用いられているPCR検査では、専門的な技術や装置を用いて検体からRNAを精製し、さらに増幅の過程を経て検出に至ります。PCR検査は感度が優れ、確定診断に適していますが、検出の前処理に最短で1時間程度がかかること、また増幅に起因する検出エラーも発生することから、大量の検体を迅速に解析し、診断につなげることが困難です。そのため、PCR検査の「感度の高さ」と抗原検査の「迅速・簡便さ」を両立させた新しいウイルス検出法の開発が急務とされてきました。

図1 従来の新型コロナウイルス感染症の診断法 抗原検査はスクリーニング用として行われ、PCR検査で診断が確定される。
2.研究手法と成果
今回、共同研究グループでは、理学と工学の異分野融合研究を推進することで、SARS-CoV-2由来のウイルスRNAを「1分子」レベルで識別して5分以内に検出する革新的技術を開発することに成功し、その手法を「CRISPR-based amplification-free digital RNA detection;SATORI)法」と名付けました。
SATORI法は、渡邉力也グループが専門とする「マイクロチップを利用した酵素反応の1分子検出技術」と西増弘志・濡木理グループが専門とする「核酸切断酵素CRISPR-Cas13a(Cas13a)」に関する先進技術を融合させたものであり、特定のRNA配列を認識するCas13aと蛍光レポーター[5]の混合液をバイオセンサーとして利用することで、検体中の標的ウイルスRNAの有無を高感度・高精度・迅速に検出できます。
SATORI法によるウイルスRNAの検出原理は以下の通りです(図2)。
- 1)核酸切断酵素Cas13aと蛍光レポーターの混合液にウイルスRNAを混ぜると、特異的にウイルスRNAとCas13aの複合体が形成されます。
- 2)複合体が形成されるとCas13aの酵素活性がオンとなり、蛍光基と消光基がつながった蛍光レポーターが切断されます。
- 3)2)の複合体と蛍光レポーターの混合液を、3フェムトリットル(fL、1 fLは1000兆分の1リットル)の微小試験管が100万個集積されたマイクロチップアレイに小分けにして封入すると、Cas13aの切断活性に伴いウイルスRNAが存在する試験管だけ蛍光シグナルが1分以内に大きく上昇します。
- 4)蛍光シグナルの有無を二値化[6]し、そのデジタル信号からシグナル有の微小試験管の個数をカウントします。カウントされる試験管の個数が検体中のウイルスRNAの個数に相当するため、ウイルスRNAの存在を1分子レベルで判別・検出できます。
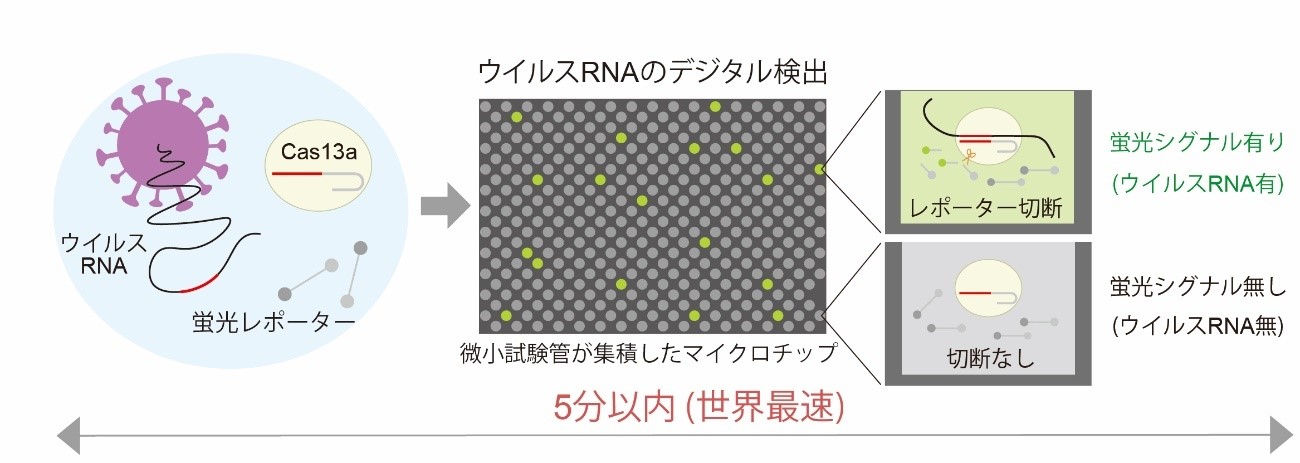
図2 SATORI法によるウイルスRNAのデジタル検出原理 核酸切断酵素Cas13aと蛍光レポーター、検体のウイルスRNAを混ぜると、特異的にウイルスRNAとCas13aの複合体が形成される。複合体が形成されるとCas13aの酵素活性がオンとなり、蛍光基と消光基がつながった蛍光レポーターが切断される。これをマイクロチップアレイに小分けにして封入すると、ウイルスRNAが存在する微小試験管だけ蛍光シグナルが1分以内に上昇する。マイクロチップの蛍光シグナルの有無を二値化し、そのデジタル信号からシグナル有の微小試験管の個数をカウントする。微小試験管内にウイルスRNAは1個だけ存在するため、カウントされる試験管の個数はサンプル中のウイルスRNAの個数に相当する。
SATORI法を用いると、5分以内にウイルスRNAを1個ずつ識別して検出できます(図3a)。検出感度は5フェムトモーラー(fM、1fMは1000兆分の1モーラー)であり、これはウイルスRNAの量で表すと、1マイクロリットル(µL、1µLは100万分の1リットル)あたり約103個となります(図3b)。この感度は従来の抗原検査法の104~105個/µLと比較すると10~100倍高く、PCR検査の10~102個/µLと比較すると10~100倍低いということになります。ただし、SARS-CoV-2感染者の検体中のウイルスRNA量は、103~106個/µLであることから、SATORI法はSARS-CoV-2の感染診断を実施する上で必要な感度を満たしているといえます。
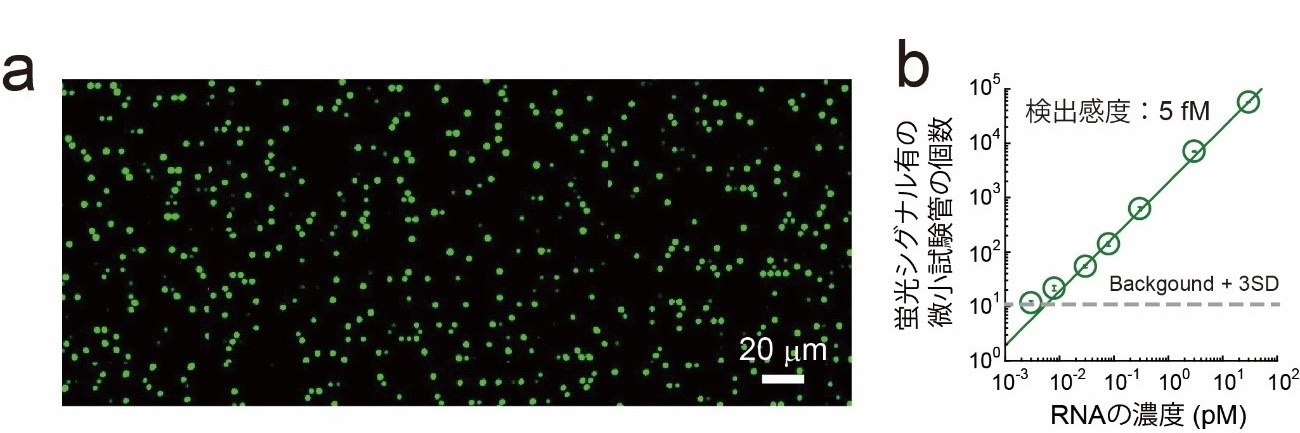
図3 SATORI法によるデジタル検出例 (a) SATORI法によるマイクロチップの蛍光画像。個々の輝点が1分子のウイルスRNAに相当する。 (b) ウイルスRNAの濃度と蛍光シグナルが確認された微小試験管の個数の相関関係。検出感度は5fM。これをウイルスRNAの量にすると、3×103個/µLとなる。
3.今後の期待
SATORI法は、ウイルスRNAを「1分子」レベルで識別して世界最高速度で検出できる革新的技術です。また、SATORI法のランニングコストはおよそ9ドルでPCR検査の5ドル程度とほぼ同等であるため、今後、消耗品の大量生産や検出装置の小型化により、安価で素早く多種のウイルス感染症を正確に診断できる次世代の感染症診断法となることが期待できます(図4)。
また、SATORI法は、疾患バイオマーカーの検出などにも活用できるため、がんなどの基礎疾患の早期・層別化診断などを指向した次世代のリキッドバイオプシー[7]の技術基盤となることも期待できます(図4)。
本研究成果は特許出願済みであり、今後は実用化を希望する企業との研究開発を進めていく予定です。
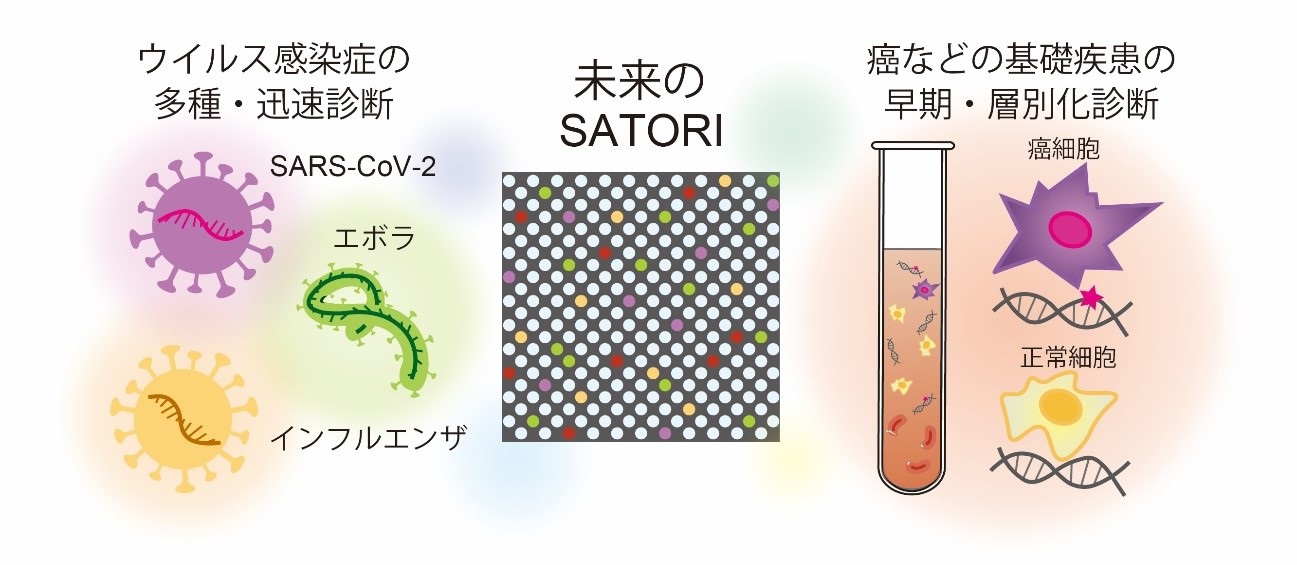
図4 リキッドバイオプシーにおけるSATORI法の将来展望 ウイルス感染症の多種・迅速診断から癌などの基礎疾患の早期・層別化診断に至る「核酸のデジタル検出」を基盤とした次世代のリキッドバイオプシーのイメージ。
4.論文情報
<タイトル>
Amplification-free RNA detection with CRISPR-Cas13
<著者名>
Hajime Shinoda, Yuya Taguchi, Ryoya Nakagawa, Asami Makino, Sae Okazaki, Masahiro Nakano, Yukiko Muramoto, Chiharu Takahashi, Ikuko Takahashi, Jun Ando, Takeshi Noda, Osamu Nureki, Hiroshi Nishimasu, Rikiya Watanabe
<雑誌>
Communications Biology
5.補足説明
- [1] PCR法
PCR法はポリメラーゼ連鎖反応法のことである。最初に、増幅対象のDNA、DNA合成酵素(DNAポリメラーゼ)、大量のプライマーと呼ばれるオリゴヌクレオチドを混合して、反応液を作る。反応液を加熱すると、2本鎖DNAが変性して1本鎖DNAになる。次に急速冷却すると、結合(アニーリング)したプライマーの3’端を起点としてDNAポリメラーゼが働き、1本鎖部分と相補的な2本鎖DNAが合成される。これで2倍量のDNAができたことになる。再び高温にしてDNA変性から繰り返す。このように、PCR法は、DNA鎖長の違いによる変性とアニーリングの違いを利用して、温度の上下を繰り返すだけでDNA合成を繰り返し、DNAを2倍、4倍、8倍、16倍…と増幅する。PCRはpolymerase chain reaction の略。 - [2] マイクロチップ技術
半導体製造プロセスを活用して微細構造をチップ上に造形する技術のこと。本研究では、容積3フェムトリットルの世界最小レベルの微小試験管を約100万個集積したマイクロチップを造形した。 - [3] 核酸切断酵素CRISPR-Cas13a
多くの細菌は、「CRISPR-Casシステム」と呼ばれる獲得免疫システムを備えている。VI型CRISPR-Casシステムに関与するCRISPR-Cas13aは、ガイドRNAと複合体を形成し、ガイドRNAと相補的な1本鎖RNAと結合すると活性化し、1本鎖RNAを切断するRNA依存性RNA切断酵素である。 - [4] 特異度
検査の性能を表す指標の一つ。陰性のものを正しく陰性と判定した割合。 - [5] 蛍光レポーター
標的RNAとCas13aの複合体を検出するための蛍光性の機能分子。核酸のウラシル(U)が五つ連なった1本鎖RNAの両端に、それぞれ蛍光基と消光基が結合した構造を持つ。複合体はウラシルが連なった構造を特異的に切断する性質を持つため、蛍光レポーターが複合体により切断されると、蛍光基は消光基から物理的に解離し、蛍光を発するようになる。 - [6] 二値化
基準となる閾値を設定し、閾値より蛍光強度が低い状態を「蛍光シグナル無(0)」、高い状態を「蛍光シグナル有(1)」として二値に変換すること。 - [7] リキッドバイオプシー
血液や尿などの身体への負担が少ない低侵襲性の液性検体の解析を基盤とした基礎疾患・感染症の診断方法。
