1.発表者:
- 森 秀人
- (慶應義塾大学大学院政策・メディア研究科 博士課程3年生)
- 石黒 宗
- (東京大学先端科学技術研究センター 合成生物学分野 特任研究員)
- 坂田 莉奈
- (東京大学教養学部国際環境学コース 学部4年生)
- 近藤 昭彦
- (神戸大学大学院科学技術イノベーション研究科 教授)
- 西田 敬二
- (神戸大学先端バイオ工学研究センター 教授)
- 冨田 勝
- (慶應義塾大学先端生命科学研究所 教授)
- 西増 弘志
- (東京大学大学院理学系研究科 生物科学専攻 准教授)
- 濡木 理
- (東京大学大学院理学系研究科 生物科学専攻 教授)
- 油谷 浩幸
- (東京大学先端科学技術研究センター ゲノムサイエンス分野 教授)
- 谷内江 望
- (東京大学先端科学技術研究センター 合成生物学分野 准教授)
2.発表のポイント:
- 細胞内の狙ったゲノムDNA配列にC→TとA→Gの同時塩基置換を誘導する新規ゲノム編集技術を開発しました
- ターゲット配列内の特定の塩基をピンポイントで置き換えることのできる塩基編集技術はこれまでにC→TまたはA→Gのいずれかを行えるものしかありませんでしたが、その両方ができるようになりました
- より高い自由度で生物のDNA配列を編集できる本ツールは品種改良、遺伝子治療、動物の発生における細胞系譜の追跡など、生物学・医学分野において幅広い応用が期待されます
3.発表概要:近年、細胞内ゲノムDNA中の特定の狙った配列を自在に編集することができるゲノム編集技術が急速に発展しており、農業、医療分野を含めた生物学分野全体に大きな変革をもたらしつつあります。これまでに、CRISPR–Cas9というゲノム編集ツールや、DNA配列を編集する塩基編集ツールが開発されてきました。これらは精密なゲノム編集技術として注目を集める一方、ゲノムに書き込まれた生命プログラムを編集するという点においては、その自由度が限られていました。慶應義塾大学院政策・メディア研究科の森 秀人 大学院生、東京大学教養学部の坂田 莉奈 学部生、東京大学先端科学技術研究センターの石黒 宗 特任研究員および谷内江 望 准教授らの研究グループは、同研究所 油谷 浩幸 教授、東京大学大学院理学系研究科 濡木 理 教授、西増 弘志 准教授、慶應義塾大学先端生命科学研究所 冨田 勝 教授、神戸大学大学院科学技術イノベーション研究科 近藤 昭彦 教授、同先端バイオ工学研究センター 西田 敬二 教授らとの共同研究により、狙ったDNA配列のC→Tおよび A→Gの異種塩基置換(注1)を同時に達成できる新たな塩基編集ツール「Target-ACEmax」の開発に成功しました。本新規ゲノム編集ツールは、さまざまな細胞においてより多様な塩基編集を可能にし、品種改良、遺伝子治療、動物の発生における細胞系譜の追跡など、さまざまな分野において幅広い応用が期待されます。
4.発表内容:生命の設計図であるゲノムはA、T、G、Cの4文字からなるDNA塩基配列で構成されています(ヒトの場合は一細胞あたり60億文字)。近年、細胞内ゲノムDNA中の特定の狙った配列を自在に編集できるゲノム編集技術が急速に発展しており、農業、医療分野を含めた生物学分野全体に大きな変革をもたらしつつあります。特に、CRISPR–Cas9ゲノム編集は、ガイドRNAというRNA分子がそれと相補的な配列を持つターゲットDNAにDNA切断活性を持つCas9タンパク質を運び、ターゲットDNAの二本鎖切断を引き起こすことができます。これが簡便に特定の遺伝子の破壊や、切断部位に別の外来DNAを挿入できるゲノム編集ツールとして広くその応用が進んできましたが、二本鎖DNA切断による細胞毒性や、編集の正確性が課題となっていました。このようななかで、この数年はDNA切断活性のない変異型のCas9に脱アミノ化酵素を融合した塩基編集ツールが開発されてきました(図1)。塩基編集ではDNAを切断せずに脱アミノ化反応を介してターゲットDNAのCをTに、あるいはAをGに高効率で変換できるため、作物の品種改良や遺伝子治療をはじめとした領域において優れたゲノム編集ツールとして期待が高まっています。しかしながら、これまでの塩基編集ツールで可能な塩基置換パターンはC→TまたはA→Gの二種類に限られていました。
今回、共同研究チームは、狙ったDNA配列においてC→Tおよび A→Gの異種塩基編集を同時に達成できる新たな塩基編集ツールTarget-ACEmaxの開発に成功しました(図2)。Target-ACEmaxはニッカーゼ型のCas9(nCas9)のN末端側にシチジン脱アミノ化酵素(注2)であるPmCDA1(C→T置換を引き起こす)を、C末端側にRNAアデノシン脱アミノ化酵素(注3)であるTadAとその変異体の二量体(A→G置換を引き起こす)が配されている他、コドン最適化(注4)および核局在シグナル(注5)の追加などによって構成されています。
本研究では、はじめに、これまでの塩基編集ツール、それらの組み合わせ、Target-ACEmaxを含め新たに開発されたいくつかの異種塩基同時編集ツールを用いて、ヒト培養細胞におけるゲノム上の47ヶ所のターゲット領域のゲノム編集を行い、これらの塩基置換パターンを超並列DNAシークエンシング(注6)によって観察しました。その結果、Target-ACEmaxが最も高い異種塩基同時編集効率があることが分かりました(図3)。また、ゲノム編集技術一般において問題視されている意図しないゲノム領域の編集(DNAオフターゲット効果、注7)や塩基編集技術で懸念されている意図しない細胞内RNA分子の編集(RNAオフターゲット効果、注8)についてエキソーム解析(注9)およびトランスクリプトーム解析(注10)を行い、Target-ACEmaxが既存の塩基編集ツールと比較していたずらにこれらのオフターゲット効果を引き上げないことも示しました(図4)。
また、超並列DNAシークエンシングによって得られた膨大な塩基編集パターン評価データを元に、任意のターゲット配列の塩基編集パターンを予測する機械学習モデルの開発にも成功しました(図5)。この機械学習モデルを用いて、Target-ACEmaxがタンパク質をコードする遺伝子群について、標的塩基群以外の不要な塩基編集を抑えて、多様なアミノ酸置換を正確に誘導できること、ヒトに観察される多様な疾患遺伝子変異の複数を同時に高効率に修復できる可能性を示唆しました。
Target-ACEmaxは、さまざまな細胞においてより多様な塩基編集を可能にし、品種改良、遺伝子治療を含めた生物学、医学分野における次世代のゲノム編集ツールとして幅広い応用が期待されます。またこの数年、谷内江准教授の研究グループでは細胞のふるまいや、細胞内分子のふるまいを経時的に細胞内の人工的なDNAのテープに記録していくような人工遺伝子回路の開発が進んでいます。この「DNAイベントレコーディング」と呼ばれる次世代の研究分野は、動物個体のそれぞれの細胞の時系列状態変化を全て記録して、後で記録された過去の状態ヒストリー情報をその個体からまるごと取り出せるような革新的技術を生み出す可能性があります。DNAイベントレコーディングにおいては、①情報センサーとしての細胞内分子回路技術、②情報記録メディアとしての人工DNA導入技術、③センサーから得た情報を人工DNAに記録する書込み技術、④人工DNAに記録された情報の読出し技術の4種類の技術コアが重要となりますが、今回開発されたDNA情報を多様に書き換えることのできるTarget-ACEmaxは③人工DNAへの情報書込み技術としても期待の高いものです。
本研究成果は2020年6月1日付けで英国科学誌「Nature Biotechnology」オンライン版に掲載されます。
論文タイトル:Base editors for simultaneous introduction of C-to-T and A-to-G mutations
著者:Rina C. Sakata†, Soh Ishiguro†, Hideto Mori†, Mamoru Tanaka, Kenji Tastuno, Hiroki Ueda, Shogo Yamamoto, Motoaki Seki, Nanami Masuyama, Keiji Nishida, Hiroshi Nishimasu, Kazuharu Arakawa, Akihiko Kondo, Osamu Nureki, Masaru Tomita, Hiroyuki Aburatani, and Nozomu Yachie* (†Equally contributed; *Corresponding author)
DOI番号:10.1038/s41587-020-0509-0
アブストラクトURL:https://www.nature.com/articles/s41587-020-0509-0
東京大学 先端科学技術研究センター 合成生物学分野
准教授 谷内江 望 (やちえ のぞむ)<報道に関すること>
東京大学 先端科学技術研究センター 広報・情報室
東京大学大学院理学系研究科・理学部 広報室
神戸大学 総務部広報課
慶應義塾大学先端生命科学研究所 渉外担当 佐藤、塩澤
DNAの異なる種類の塩基を同時に変換することの意。(注2)シチジン脱アミノ化酵素:
DNAのシチジン(C)からアミノ基を除き、ウリジン(U)に変換するタンパク質。UはDNA複製時にTに変換される。(注3)RNAアデノシン脱アミノ化酵素:
RNAのアデノシン(A)からアミノ基を除き、イノシン(I)に変換するタンパク質。本研究ではDNAのAをIに変換するこれの改変体タンパク質が用いられた。IはDNA複製時にGに変換される。(注4)コドン最適化:
対象とするタンパク質が対象とする細胞内で効率良く発現するように遺伝子暗号コードを最適化することの意。(注5)核局在シグナル:
核内へ局在が誘導されるタンパク質ドメイン。これを付加することで効率良く染色体を格納する核内へゲノム編集ツールなどを導入することができる。(注6)超並列DNAシークエンシング:
次世代DNAシークエンシング。超並列に数千万~数十億分子の短いDNA配列を同時に解読できる。(注7)DNAオフターゲット効果:
ゲノム編集が意図しないDNA領域を編集してしまう効果。(注8)RNAオフターゲット効果:
ゲノム編集が細胞中の意図しないRNA分子を編集してしまう効果。(注9)エキソーム解析:
ゲノム中のタンパク質コード領域のDNA変異を一斉に超並列DNAシークエンシングで一斉に計測する技術。
(注10)トランスクリプトーム解析:
細胞内のRNA分子配列を一斉に計測する技術。
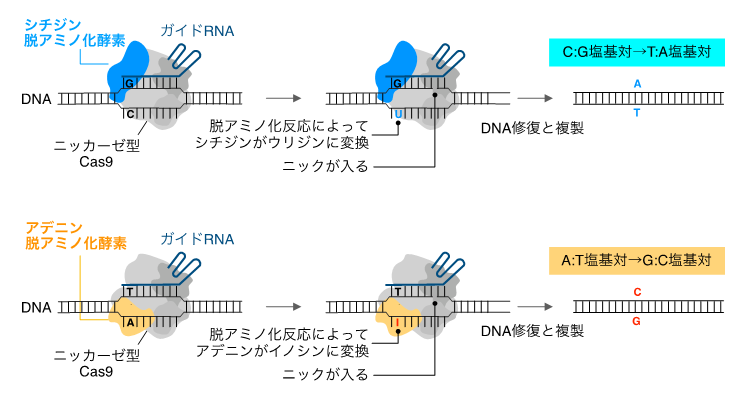
図1 従来の塩基編集技術。ニッカーゼ型Cas9がシチジンまたはアデニン脱アミノ化酵素を持っており、それぞれC→T置換あるいはA→G置換を誘導できる。
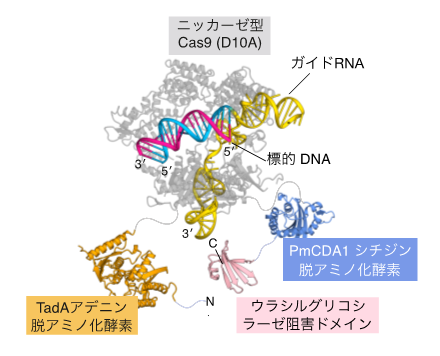
図2 Target-ACEmax。PmCDA1シチジン脱アミノ化酵素およびTadAアデニン脱アミノ化酵素の両方を持ち、DNAのターゲット配列にC→T置換およびA→G置換を同時に誘導できる。
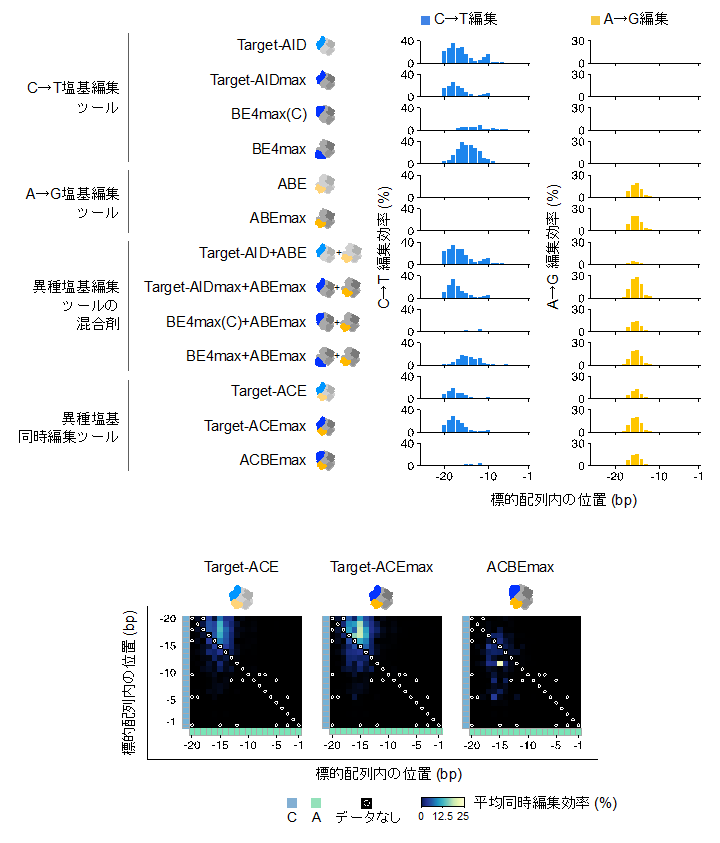
図3 さまざまな塩基編集ツールによってヒトゲノム標的サイトを編集したときの平均編集スペクトラムと異種塩基同時編集頻度。単体の塩基編集ツールとしてはTarget-ACEmaxが最も高い異種塩基同時編集頻度を示した。
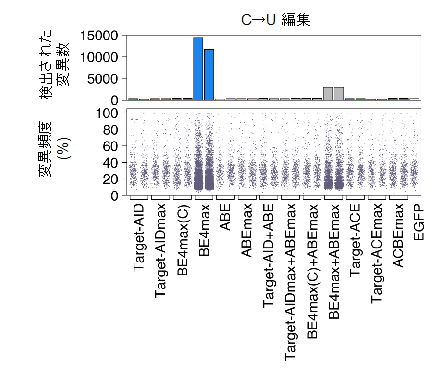
図4 さまざまな塩基編集ツールのRNAオフターゲット効果によって引き起こされるヒト細胞内RNA分子のC→U変異。観測されたRNA変異総数と各RNA変異の頻度。Target-ACEmaxのRNAオフターゲット効果は低い。
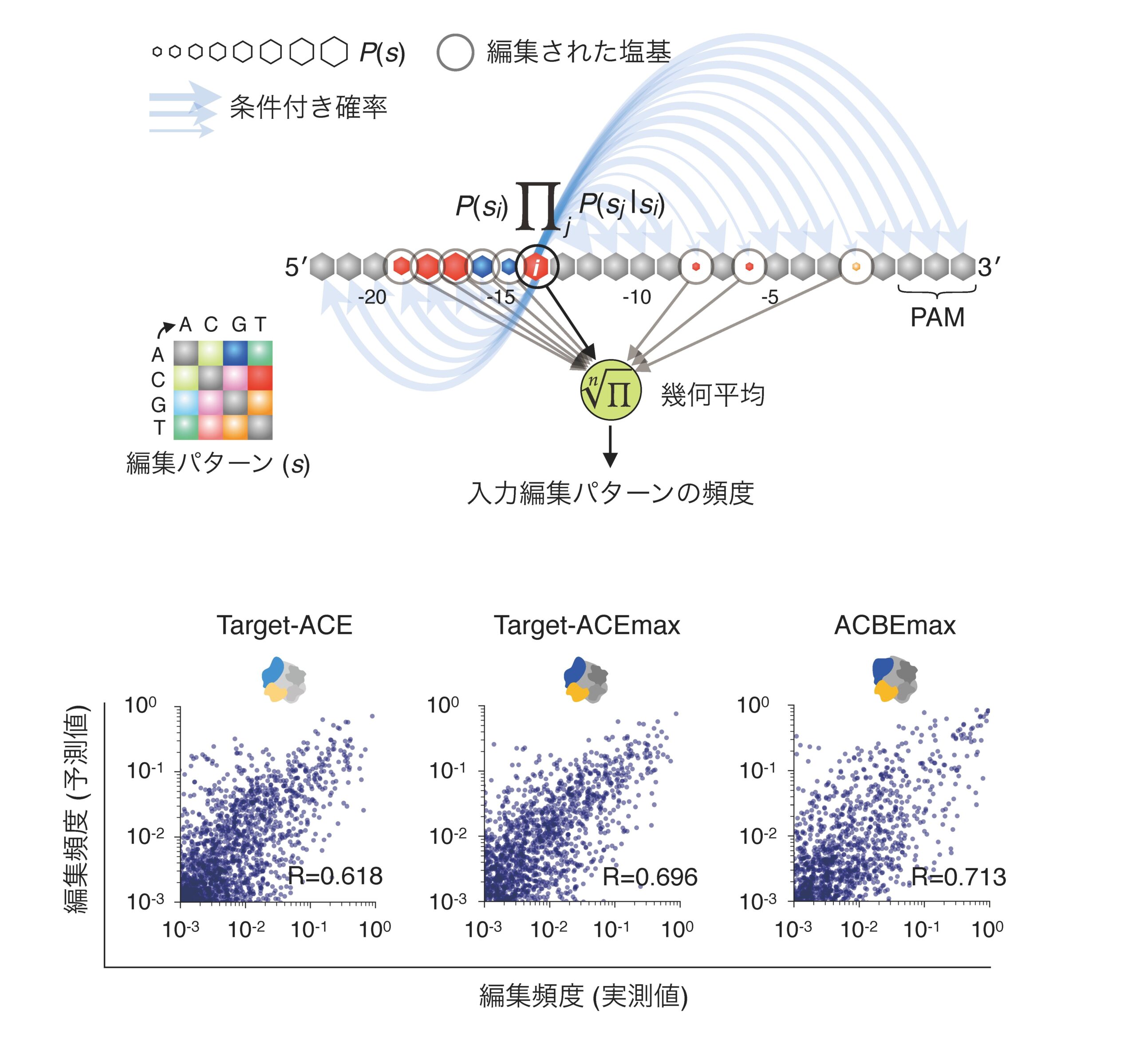
図5 条件付き確率モデルによる異なる塩基編集ツールによる任意のターゲットDNA配列のさまざまな塩基編集パターン頻度の予測。さまざまな塩基編集ツールについてゲノム編集結果の予測が実測値と概ね似たものになる。
